1er Foro Internacional de Bioinformática:
Ciencia y Tecnología para el futuro del país
Silvia y Marcela
Jordan-Bogota-April2014
Eduardo Vela
Carlos Vasquez
Diego Riano UA_MBC_DMRP_2014
Marco Cristancho
Jorge Duitama
BIOS Official Spanish Apr2014 - UniAndes
Maria Mercedes
Alejandro Delgado
Fernando Barraza
e-Clouds A Platform and Marketplace to Access and Publish Scientific Applications as a Service - Foro Bioinformática - Mario Villamizar
Video Introductorio
Videos de Panelistas
Silvia Restrepo, decana de la Facultad de Ciencias de la Universidad de los Andes, interviene durante el 1er Foro Internacional de Bioinformática, organizado por el Departamento de Ingeniería de Sistemas y Computación de Uniandes.
Jorge Duitama, investigador en bioinformática del Centro Internacional de Agricultura Tropical, interviene durante el 1er Foro Internacional de Bioinformática, organizado por el Departamento de Ingeniería de Sistemas y Computación de Uniandes.
Mauricio Rodríguez, director general del Centro Colombiano de Bioinformática y Biología Computacional (BIOS), interviene durante el 1er Foro Internacional de Bioinformática, organizado por el Departamento de Ingeniería de Sistemas y Computación de Uniandes
Mario Villamizar, profesor del Departamento de Ingeniería de Sistemas y Computación de la Universidad de los Andes, interviene durante el 1er Foro Internacional de Bioinformática, organizado por Uniandes.
Marco Cristancho, investigador científico de Cenicafé, interviene durante el 1er Foro Internacional de Bioinformática, organizado por el Departamento de Ingeniería de Sistemas y Computación de Uniandes.
King Jordan, profesor asociado de Georgia Tech (Estados Unidos), interviene durante el 1er Foro Internacional de Bioinformática, organizado por el Departamento de Ingeniería de Sistemas y Computación de Uniandes.
Diego Riaño, representante del Laboratorio Nacional de Ciência e Tecnologia do Bioetanol (CTBE), interviene durante el 1er Foro Internacional de Bioinformática, organizado por el Departamento de Ingeniería de Sistemas y Computación de Uniandes
Fernando Barraza, líder I+D de Parquesoft, interviene durante el 1er Foro Internacional de Bioinformática, organizado por el Departamento de Ingeniería de Sistemas y Computación de Uniandes.
Alonso Verdugo, client technical professional -- software industry solutions -- i2 portfolio de IBM, interviene durante el 1er Foro Internacional de Bioinformática, organizado por el Departamento de Ingeniería de Sistemas y Computación de Uniandes.

Con el patrocinio de:
![]()
Con la participanción de:
![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]()



Media partner:
![]()
Objetivo
El mundo está de lleno en la nueva era de la Biología Computacional. Esta rama de la Biología y de las Ciencias Computacionales tiene un rol esencial en estudios a gran escala en temas que van desde la biodiversidad y la conservación hasta la solución de problemas en salud y agricultura que pueden significar la supervivencia humana en el mundo. Cada día se producen en el mundo millones de datos en el estudio de la Biología y la Medicina y gracias a los avances computacionales es ahora factible realizar análisis que hace unos años no habrían sido posibles.
La generación de estas cantidades masivas de datos es cada vez más económica y asequible. Sin embargo, el factor limitante ahora es el personal calificado y entrenado con la capacidad de analizar e interpretar los datos. Colombia debe posicionarse en esta área en la que tiene un alto potencial de ser competitiva. En este foro se discutirán tecnologías de punta en Biología Computacional, así como el rol que juegan para acelerar el desarrollo del país.
Fecha:
Jueves 10 de Abril 2014
Hora:
7:30 A.M. - 7:00 P.M.
Lugar:
Universidad de los Andes- Auditorio A y B del Edificio Mario Laserna. Cra. 1 Este No. 19 A - 40
| Hora | Evento |
| 7:00 a.m. - 8:00 a.m. | Registro |
| 8:00 a.m. - 9:00 a.m. | Introducción José Rafael Toro, Vicerrector Académico Uniandes Silvia Restrepo, Decana Facultad de Ciencias Uniandes Marcela Hernández, Profesora Ingeniería de Sistemas y Computación Uniandes |
| Track Visión Internacional | |
| 9:00 a.m. - 9:40 a.m. | Charla: Ancestry, Admixture and Selection in Colombian Genomes King Jordan, Georgia Tech |
| 9:40 a.m. - 10:10 a.m. | Charla: Nuevas Capacidades para un Nuevo País Carlos Vásquez, Microsoft |
| 10:10 a.m. - 10:40 a.m. | Charla: Global Technology Outlook 2014 Alonso Verdugo, IBM |
| 10:40 a.m. - 11:00 a.m. | Café |
| Track científico | |
| 11:00 a.m. - 11:20 a.m. | Charla: Estudios genómicos de especies relevantes en la cadena productiva del bioetanol en Brasil Diego Riaño, CTBE |
| 11:20 a.m. - 11:40 a.m. | Charla: Diferenciando el Café de Colombia a través de estudios de Genómica y Bioinformática Marco Cristancho, Cenicafé |
| 11:40 a.m. - 12:00 m | Charla: Genética de poblaciones y mejoramiento de cultivos utilizando tecnologías de secuenciación de alto rendimiento Jorge Duitama, CIAT |
| 12:00 m. - 2:00 p.m. | Almuerzo |
| Track Empresarial | |
| 2:00 p.m. - 2:20 p.m. | Charla: Hacia la bioeconomía basada en el conocimiento a través de la alta tecnología Mauricio Rodríguez, BIOS |
| 2:20 p.m. - 2:40 p.m. | Charla: Corpogen: adaptación en la era digital María Mercedes Zambrano, CORPOGEN |
| 2:40 p.m. - 3:00 p.m. | Charla: La transformación productiva a través de la bioinformática Alejandro Delgado, PTP |
| 3:00 p.m. - 3:20 p.m. | Charla: Servicios en la nube para proyectos en Bioinformática: consideraciones técnicas y de negocio Fernando Barraza, Parquesof |
| Track Técnico | |
| 3:20 p.m. - 3:45 p.m. | Charla: Cloud Científico Mario Villamizar, Uniandes |
| >3:45 p.m. - 4:10 p.m. | Café |
| Panel de Discusión | |
| 4:10 p.m. - 5:30 p.m. | "La bioinformática en Colombia" |
| 5:30 p.m. - 7:00 p.m. | Coctel y Sesión de Networking |
Panelistas

Alonso Verdugo
Actualmente, Alonso trabaja en IBM (Client Technical Professional – Software Industry Solutions – i2 Portfolio). La tecnología i2 permite a las agencias gubernamentales y empresas del sector privado investigar, predecir, prevenir y detener las amenazas criminales y terroristas más sofisticadas del mundo. Alonso enseña a los clientes cómo utilizar esta tecnología para su negocio (Seguridad del hogar y de los territorios, Policía, Finanzas, Retail, Seguros, Telecomunicaciones, Salud, etc) Sus certificaciones incluyen IBM SOA Design, COBIT e ITIL.

Carlos Vásquez
Carlos Vásquez, Ingeniero de Sistemas con 26 años de experiencia laboral e investigativa tanto en Colombia, Latinoamérica y EE.UU. Desde hace 18 años trabaja en Microsoft en diferentes posiciones tanto locales como Regionales. Nueve (9) años en la división de Servicios Empresariales de Consultoría y ha participado en el desarrollo de productos en la casa matriz de Microsoft en Seattle, EE.UU.
Actualmente se desempeña en Microsoft como Director Nacional de Tecnología (National Technology Officer (NTO)) para Colombia, quien actúa como vocero líder para la subsidiaria en todos los aspectos de estrategia tecnológica de la compañía a futuro y su relación con el desarrollo de políticas públicas para el uso de la tecnología en Gobierno, y Academia así como en proyectos sociales del país.

King Jordan
King Jordan es Profesor Asociado de la Escuela de Biología y Director del Programa de Postgrado en Bioinformática en el Instituto de Tecnología de Georgia. Obtuvo un doctorado en Genética de la Universidad de Georgia en 1998 y posteriormente se incorporó como investigador postdoctoral en el Departamento de Ciencias Biológicas de la Universidad de Nevada. Desde 2002 hasta 2006 estuvo trabajando en el Centro Nacional de Información sobre Biotecnología-NCBI en diferentes posiciones, y luego comenzó a trabajar en la tecnología de Georgia.
Sus principales intereses son el desarrollo y la aplicación de algoritmos para el análisis genómico funcional de alto rendimiento de datos de secuencia: montaje del genoma, ChiP-seq, RNA-seq y la creación de programas de la web habilitada para la epidemiología molecular y la genómica comparativa.

Diego Riaño
Entre 2010 y 2012 el Dr. Riaño-Pachón fue profesor Asistente en el Departamento de Ciencias Biológicas de la Universidad de Los Andes en Bogotá, donde lideró el grupo de investigación en Biología Computacional y Evolutiva, integrado por estudiantes de pregrado y postgrado. Desde Febrero de 2013, dirige un grupo de investigación en el Laboratorio Nacional de Ciencia y Tecnología del Bioetanol (CTBE), parte del Centro Nacional de Investigación en Energía y Materiales (CNPEM), en Campinas, São Paulo, Brasil (http://bce.bioetanol.cnpem.br/)

Marco Cristancho
Microbiólogo de la Universidad de los Andes, con PhD en Biología Molecular de Plantas de la Universidad de Manchester y Postdoctorado en Bioinformática de Cornell University. Actualmente, es Investigador de Cenicafé, lidera proyectos para la caracterización y el control de la roya del café, es miembro del equipo de trabajo de la disciplina de Fitopatología, y líder del grupo de Bioinformática del Centro.

Jorge Duitama
Ingeniero y Magister en ingeniería de sistemas de la Universidad de los Andes. Ph.D. en ciencias de computación de la Universidad de Connecticut. Trabajó en el Instituto Max Planck de genética molecular como asistente de investigación y en la Universidad Católica de Lovaina como postdoc. Actualmente trabaja como investigador en bioinformática en el CIAT en donde investiga el uso de las tecnologías de secuenciación de alto rendimiento como herramienta para el desarrollo de variedades mejoradas de arroz, yuca, frijol y pastos.

Mauricio Rodriguez
El Dr. Mauricio Rodríguez es actualmente Director General del Centro de Bioinformática y Biología Computacional de Colombia, también conocida como BIOS. Anteriormente se desempeñó como Gerente del Programa Nacional de Biotecnología, en el Departamento Administrativo de Ciencia, Tecnología e Innovación – COLCIENCIAS.

María Mercedes Zambrano
Bióloga de Wellesley College (EEUU) y PhD en Microbiología y Genética Molecular de la Universidad de Harvard (EEUU). Fundamos la Corporación Corpogen con la Dra. Patricia Del Portillo en 1995, donde he trabajado desde entonces. En el momento soy Directora Científica, superviso varias tesis y lidero proyectos de investigación en las áreas de la Ecología microbiana, metagenómica y patogénesis bacteriana. En el momento también lidero el Centro de Genómica y Ambientes Extremos Gebix, una red de trabajo en metagenómica y bioprospección.

Alejandro Delgado
Gerente del sector de software y Ti del programa de transformación productiva del Ministerio de comercio, industria y turismo. Administrador de empresas de la Universidad de Louisiana y MBA de la misma universidad, especialista de Geopolítica de la universidad EAFIT, maestría de análisis de problemas económicos y políticos internacionales de la academia diplomática colombiana.

Fernando Barraza
Profesor Asociado, Coordinador Maestría en Ingeniería de Software Universidad de San Buenaventura Cali.

Mario Villamizar
Experto temático en proyectos de Cloud Computing y docente de la Universidad de los Andes donde participa, investiga y lidera proyectos a nivel nacional e internacional relacionados con modelos de negocio en Cloud Computing, gestión y adopción de tecnologías Cloud a nivel empresarial, diseño de infraestructura de TI escalables y automatizadas, desarrollo ágil de aplicaciones web/móviles para plataformas Cloud e implementación de soluciones de Software como servicio.


Flujos de Trabajo de Bioinformatica en la Nube
Análisis de secuencias al alcance de todos
¿A quién está dirigido?
Este taller teórico-práctico está dirigido a investigadores en ciencias biológicas, ciencias de la computación y áreas afines que estén interesados en el análisis de secuencias (que no requieren uso de la línea de comandos), las cuales explotan el poder computacional disponible en la nube, con servidores gratuitos o que pueden ser alquilados de acuerdo a las necesidades de los grupos de investigación.
¿Qué temáticas se tratarán?
El taller inicia con una introducción al proyecto Galaxy, el cual, mediante una interfaz web, permite no solo llevar análisis individuales de datos biológicos, sino también inter-conectarlos para formar flujos de trabajo complejos, en una forma dinámica y amigable con el usuario.
La segunda parte del taller, dará una introducción a la computación en la nube, familiarizando al participante con las diferentes opciones y herramientas disponibles. En particular, se mostrará una aproximación práctica al uso del BLAST en la nube resaltando las ventajas y desventajas de esta aproximación.
Costo
Estudiantes*:$ 100.000
Externos:$ 250.000
*Los estudiantes deben presentar carnet vigente para ingresar al taller
Lugar
Salas Waira (Laboratorios ML 612 y 613)
Edificio Mario Laserna, Universidad de los Andes
Cra 1Este No. 19 A – 40
Fechay hora
Fecha: 9 de abril
Hora: 5:00 p.m. a 9:00 p.m.
contacto
María Isabel Rodriguez
mi.rodriguez21-at-uniandes.edu.co
Dictado por

Diego Riaño
- Postdoctorado en Bioinformática del Instituto Max Plank de Fisiología Molecular Vegetal, Universidad de Potsdam, Alemania
- Ph.D.en Biología Molecular Vegetal, Universidad de Potsdam, Alemania
- Biólogo, Universidad Nacional de Colombia
Entre 2010 y 2012 el Dr. Riaño-Pachón fue profesor Asistente en el Departamento de Ciencias Biológicas de la Universidad de Los Andes en Bogotá, donde lideró el grupo de investigación en Biología Computacional y Evolutiva, integrado por estudiantes de pregrado y postgrado. Desde Febrero de 2013, dirige un grupo de investigación en el Laboratorio Nacional de Ciencia y Tecnología del Bioetanol (CTBE), parte del Centro Nacional de Investigación en Energía y Materiales (CNPEM), en Campinas, São Paulo, Brasil (http://bce.bioetanol.cnpem.br/).
Ha publicado más de 20 artículos de investigación, que han recibido más de 1900 citaciones en total. Sus principales líneas de actuación incluyen el desarrollo de bases de datos para la integración de datos provenientes de tecnologías ómicas, los métodos de clasificación y aprendizaje de máquina aplicados a problemas biológicos, el estudio de la regulación de la transcripción en eucariotes, la evolución de las redes de regulación génica, y el ensamblaje y anotación de genomas de diversos procariotes y eucariotes. Entre sus intereses también está el desarrollo de flujos de trabajo y el despliegue de herramientas que faciliten el uso de infraestructura computacional de alto rendimiento.
Mario Villamizar
- Master en Ingeniería de Sistemas y Computación, Universidad de los Andes
- Ingeniero de Sistemas, Universidad Francisco de Paula Santandar
Experto temático en proyectos de Cloud Computing y docente de la Universidad de los Andes donde participa, investiga y lidera proyectos a nivel nacional e internacional relacionados con modelos de negocio en Cloud Computing, gestión y adopción de tecnologías Cloud a nivel empresarial, diseño de infraestructura de TI escalables y automatizadas, desarrollo ágil de aplicaciones web/móviles para plataformas Cloud e implementación de soluciones de Software como servicio.
Mario ha sido autor de más de 30 publicaciones internacionales en temas relacionados con computación de alto rendimiento, Grid y Cloud Computing. Sus áreas de interés actuales son Cloud Computing arquitecturas de aplicaciones escalables y proyectos de emprendimiento en internet.
3er Foro Internacional de Biología Computacional:

El mundo está de lleno en la nueva era de la Biología Computacional. Esta rama de la Biología y de las Ciencias Computacionales tiene un rol esencial en estudios a gran escala en temas que van desde la biodiversidad y la conservación hasta la solución de problemas en salud y agricultura que pueden significar la supervivencia humana en el mundo. Cada día se producen en el mundo millones de datos en el estudio de la Biología y la Medicina y gracias a los avances computacionales es ahora factible realizar análisis que hace unos años no habrían sido posibles.
La generación de estas cantidades masivas de datos es cada vez más económica y asequible. Sin embargo, el factor limitante ahora es el personal calificado y entrenado con la capacidad de analizar e interpretar los datos. Colombia debe posicionarse en esta área en la que tiene un alto potencial de ser competitiva. En este foro se tendrán charlas de importantes expertos internacionales en el área de la metagenómica y la caracterización de comunidades y diversidad ambiental. Se complementarán con las presentaciones y defensas de tesis de los estudiantes de la Maestría en Biología Computacional así como charlas cortas de estudiantes e invitados.
*Cambios sin previo aviso
Panelistas
Daniel McDonald - University of California San Diego
Charla: Rosetta and the expansion of the American Gut Project
Position: Scientific Director, American Gut Project
Affiliation: Department of Pediatrics, School of Medicine, UC San Diego
Dr. McDonald is the Scientific Director for The Microsetta Initiative and the American Gut Project, run by the School of Medicine at UC San Diego. His research focuses on the complex microbial communities associated with humans, with the environment, and how to scale microbiome analysis to large sample sizes. He been heavily involved in the development of popular computational tools, such as QIIME, Qiita, and PICRUSt, and redeveloped the commonly used phylogenetic algorithm, UniFrac, to support hundreds of thousands of microbiome samples. His publication record in human associated studies includes population scale assessments, examining treatment naive inflammatory bowel disease patients, forensic uses of the microbiome, the identification of novel dietary parameters associated with microbial composition, and integration of multi-omic data. Dr. McDonald received a BS and PhD in Computer Science from the University of Colorado at Boulder.
Twitter, LinkedIn (if desired): @mcdonadt
Bent Petersen
Charla: Examining the diversity of the Amazon rainforest
Position: Associate Professor
Affiliation: Natural History museum of Denmark, Copenhagen University
Bent Petersen is an Associate Professor in bioinformatics at the Natural History museum of Denmark, Copenhagen University. He is also the Deputy Director for the Centre of Excellence for Omics-Driven Computational Biodiscovery (COMBio) at AIMST University, Malaysia. He has a thorough experience in machine learning and has further specialized within the field of genomics and metagenomics where he is working extensively with Next Generation Sequencing data. His research group actively collects samples from all over the world and develops new tools that address the unique challenges of microbial and antique systems biology often by using machine learning.
Currently, he is working extensively on genome and metagenome projects from various branches of the tree of life where they are using supercomputers to analyse the data. In the recent years, drawn by the challenges of plant genome data analysis, Bent has further developed in the field of plant genomics and established the Amazon Rainforest Genome Ontology project (ARGO). A website with video of the project can be seen here: http://www.amazonia.life/.
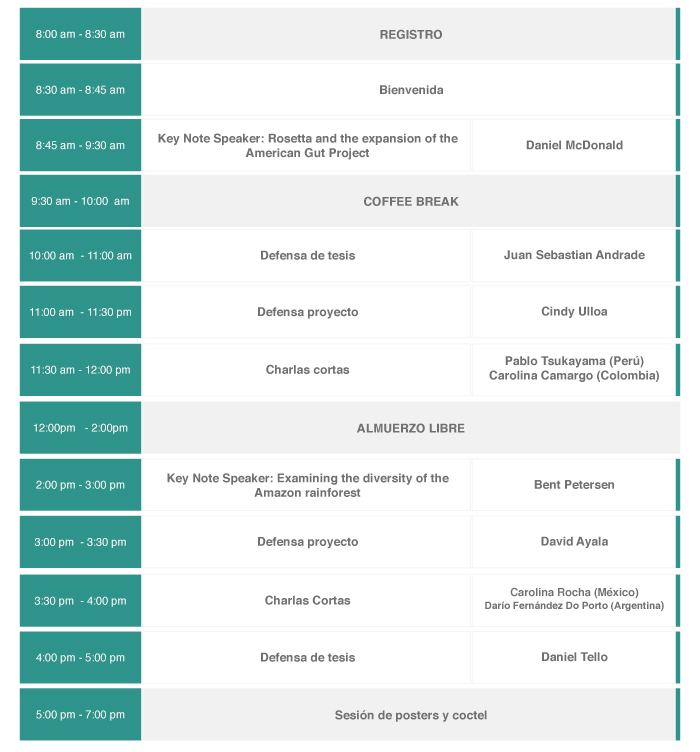
6to Congreso Colombiano de Biología Computacional Bioinformática

La semana del 28 de marzo al primero de abril de 2022 se llevó a cabo en la sede Caribe de Cartagena de la Universidad de los Andes la sexta versión del congreso Colombiano de Bioinformática y Biología Computacional de Colombia. Este evento contó con la participación de más de cien investigadores, empresarios y estudiantes interesados en las diferentes áreas de conocimiento que hoy en día tienen relación con la bioinformática.
Durante los dos primeros días se realizaron dos cursos precongreso, el primero en bioinformática estructural y el segundo en ensamblaje de genomas y análisis de datos poblacionales. Los cursos fueron tomados por más de 20 estudiantes interesados en los dos temas. El curso de bioinformática estructural contó con el apoyo de una de las invitadas internacionales, la profesora Ezgi Karaka del centro Izmir de biomedicina y genómica de Turquía. Durante estos dos días también se llevó a cabo el evento de estudiantes latinoamericanos de la sociedad internacional de biología computacional (ISCB).
Durante los tres días siguientes los asistentes tuvieron la oportunidad de compartir y discutir los diferentes temas de investigación básica y aplicada propuestos por el programa, a través de 17 charlas invitadas y 46 charlas de estudiantes. El primer día se dió énfasis en bioinformática aplicada a mejoramiento de cultivos. El profesor Alfredo Herrera de CINVESTAV dió inicio a este día con su charla sobre genómica para acelerar la caracterización y mejoramiento de cultivos estratégicos en Mexico. Este grupo de charlas incluyó también en la sesión de la mañana la charla de Máximo Rivarola del INTA sobre la integración de bioinformática al mejoramiento de cultivos en Argentina y de la investigadora Paula Reyes de Agrosavia sobre la bioinformática como una aliada para el sector agropecuario Colombiano. Esta sesión continuó por la tarde con las charlas del investigador Marco Cristancho de la Universidad de los Andes sobre genómica de patógenos que afectan cultivos en latinoamérica y del profesor Edison Chavarro de la Universidad Tecnológica de Bolívar sobre genómica de Rhizoctonia solani. Finalmente, se cerró el día con la charla del investigador Jan Kreuze del Centro Internacional de la Papa (CIP) sobre genómica en virología de cultivos.
De manera paralela se dio inicio a la sesión de biología computacional y COVID con la charla de la investigadora Marcela Guevara de la Universidad de los Andes sobre la experiencia en genómica para diagnóstico de COVID en Colombia. Por su parte, el profesor Yesid Cuesta del Instituto Colombiano de Medicina Tropical sobre el “proyecto Jaguar”, con el cual se espera utilizar tecnología de secuenciación de ARN de célula sencilla para mapear la diversidad de células inmunes. En la tarde se llevó a cabo la sesión de bioinformática y enfermedades infecciosas, la cual incluyó las charlas de Adrian Turjansky de la Universidad de Buenos Aires sobre genómica para desarrollo de drogas contra diferentes patógenos humanos y la charla del profesor Daniel Urrea sobre arquitectura genómica de tripanosomátidos. Finalmente, el profesor Guillerme Oliveira del Instituto tecnológico Vale sobre genómica y bioinformática aplicada a la conservación de la biodiversidad en el Amazonas.
El segundo día de charlas inició con la charla invitada de la investigadora Natasa Przulj del centro de supercomputación de Barcelona sobre modelos de algorítmica e inteligencia artifical para integración de datos heterogéneos en medicina de precisión. Con esto se dió inicio a la sesión de genética humana, la cual fue apoyada por la charla de la investigadora Silvia Maradei de Biotecgen sobre secuenciación de genomas humanos para diagnóstico genético y del investigador Narmer Galeano del BIOS sobre el “Programa origen” para implementar la medicina de precisión en Colombia. Por otra parte, se llevó a cabo la sesión de Ecología Microbiana, la cual fue iniciada por la charla del profesor Alejandro Reyes de la Universidad de los Andes sobre el uso de bases de datos públicas para descifrar la materia oscura viral.
Durante la tarde se llevaron a cabo las sesiones de genómica, transcriptómica y proteómica funcional y de biología de sistemas. En estas dos sesiones se destacan las charlas de la profesora Liliana Lopez Kleine de la Universidad Nacional de Colombia sobre detección de genes diferencialmente expresados en datos de RNA-seq de célula sencilla y del profesor Andres Gonzalez de la Universidad de los Andes sobre biología de sistemas en la solución de problemas de la industria Colombiana. El segundo día se cerró con la charla de la profesora invitada Ezgi Karaka del centro Izmir de biomedicina y genómica de Turquía sobre detección de sitios de interacción molecular.
Finalmente el viernes se llevaron a cabo las sesiones de desarrollo de software para bioinformática y de modelamiento molecular. De estas sesiones se destacan las charlas del profesor Jorge Duitama de la Universidad de los Andes sobre desarrollo de software para genómica evolutiva y de poblaciones, la del profesor Carlos Muskus de la Universidad de Antioquia sobre análisis de ARN ribosomales en parásitos de Leishmania resistentes a antimoniales y la del profesor Juan Carlos Cruz sobre ingeniería de péptidos para terapias génicas dirigidas.
Estas charlas permitieron guiar la discusión esperada alrededor de la gran variedad de temas propuestos en el congreso, abarcando temas importantes en ciencias básicas como la evolución de especies, dinámica molecular y biología de sistemas, así como diferentes aplicaciones en medicina, conservación, producción de alimentos y bioeconomía. Además de las charlas, tuvimos la exposición de 30 posters de investigadores de diferentes instituciones.
Tuvimos 120 asistentes de diferentes instituciones educativas, centros de investigación y empresas. Participaron investigadores y estudiantes de 21 universidades localizadas en nueve departamentos de Colombia (Antioquia, Atlántico, Bogotá, Bolívar, Boyacá, Norte de Santander, Quindío, Santander y Valle). Participaron también investigadores de centros de investigación públicos y privados de Colombia, incluyendo Agrosavia, BIOS, Cenicaña, Cenipalma, Corpogen, la Corporación para Investigaciones Biológicas y el Instituto Colombiano de Medicina Tropical. Asimismo, participaron delegados de entidades del sector público y privado, incluyendo Biotecgen S.A, Sanitas, el ICA y el Instituto Nacional de Cancerología. Finalmente, tuvimos la participación de investigadores y estudiantes de siete Universidades extranjeras quienes nos acompañaron desde países como Brasil, Chile, Estados Unidos, México y Perú.
Este congreso no hubiera podido ser exitoso sin haber contado con el apoyo de nuestros patrocinadores Biotecgen S.A, Corpogen, y la maestría en biología computacional de la Universidad de los Andes. En nombre de los organizadores: Universidad de los Andes, Universidad de Antioquia, el Instituto Colombiano de Medicina Tropical, el proyecto CABANA, la Universidad del Tolima, la Universidad tecnológica de Bolívar, la Sociedad Internacional de Biología Computacional (ISCB), la Universidad Nacional de Colombia, Agrosavia y BIOS, les agradecemos nuevamente por su participación en CCBCOL VI y los invitamos a participar en la próxima edición del congreso, a realizarse en Medellín en 2024.












2do Foro Internacional de Biología Computacional:

Apoyado por:

Objetivo
El mundo está de lleno en la nueva era de la Biología Computacional. Esta rama de la Biología y de las Ciencias Computacionales tiene un rol esencial en estudios a gran escala en temas que van desde la biodiversidad y la conservación hasta la solución de problemas en salud y agricultura que pueden significar la supervivencia humana en el mundo. Cada día se producen en el mundo millones de datos en el estudio de la Biología y la Medicina y gracias a los avances computacionales es ahora factible realizar análisis que hace unos años no habrían sido posibles.
La generación de estas cantidades masivas de datos es cada vez más económica y asequible. Sin embargo, el factor limitante ahora es el personal calificado y entrenado con la capacidad de analizar e interpretar los datos. Colombia debe posicionarse en esta área en la que tiene un alto potencial de ser competitiva. En este foro se discutirán tecnologías de punta en Biología Computacional, así como el rol que juegan para acelerar el desarrollo del país.
Igualmente, este foro es un día para que los estudiantes que se encuentran trabajando en el área de Biología Computacional y Bioinformática compartan sus trabajos con compañeros y profesores.
Fecha:
Viernes 1 de Diciembre 2017
Hora:
8:00 A.M. - 5:00 P.M.
Inscripción previa
Inscripciones abiertas hasta el 29 de noviembre. Cupos limitados
Lugar:
Salón ML 603
Universidad de los Andes. Edificio Mario Laserna.
*Cambios sin previo aviso
| Hora | Evento |
| 7:30 a.m. - 8:00 a.m. | Registro |
| 8:00 a.m. - 8:10 a.m. | Bienvenida |
| 8:10 a.m. - 9:00 a.m. | Keynote Marco Vignuzzi |
| 9:00 a.m. - 9:30 a.m. | Marco Cristancho: CABANA poject |
| 9:30 a.m. - 10:00 a.m. | Presentación trabajo grado (Camila Martinez) |
| 10:00 a.m. - 10:15 a.m. | Refrigerio |
| 10:15 a.m. - 12:00 p.m. | Charlas cortas (7 charlas) |
| 12:00 p.m. - 2:00 p.m. | Almuerzo libre |
| 2:00 p.m. - 2:30 p.m. | Charla Investigación (Claudia Chica) |
| 2:30 p.m. - 3:15 p.m. | Charlas Cortas (3 charlas) |
| 3:15 p.m. - 4:15 p.m. | Presentación trabajo grado (Manuela Vanegas) |
| 4:15 p.m. - 4:30 p.m. | Refrigerio |
| 4:30 p.m. - 6:00 p.m. | Sesión de posters |
| 6:00 p.m. - 8:00 p.m. | Cóctel/Posters |
Panelistas

Marco Vignuzzi
Principal Investigator, of the Viral Populations and Pathogenesis Unit at Institut Pasteur. His lab tests concepts in evolution within the field of virology. They use cutting edge technologies and develop computational and experimental approaches to monitor, and potentially predict, virus evolution. This knowledge is then used in new vaccine and antiviral approaches.
Marco did his B.Sc. in Microbiology and Immunology at the McGill University in Montreal Canada. He then moved to France where he did hid Masters in Fundamental Virology at the University of Paris – Denis Diderot an his Ph.D., 2001 in Virology under the supervision of Sylvie van der Werf at the Institut Pasteur. After his Postdoct at the University of California, San Francisco in Raul Andino’s Lab he came back to the Institut Pasteur to lead his own lab.
Hands-on course on High Throughput Sequencing data analysis
Genomics, Transcriptomics, Epigenomics
Apoyado por:

¿A quién está dirigido?
Este taller teórico-práctico está dirigido a investigadores en ciencias biológicas, ciencias de la computación y áreas afines que estén interesados en el análisis de secuencias (que no requieren uso de la línea de comandos), las cuales explotan el poder computacional disponible en la nube, con servidores gratuitos o que pueden ser alquilados de acuerdo a las necesidades de los grupos de investigación.
¿Qué temáticas se tratarán?
The University of Los Andes and the Hub of Bioinformatics and Biostatistics of the C3BI (Center of Bioinformatics Biostatistics and Integrative Biology) at Institut Pasteur will organize this Hands-on course aimed at students/researchers that have produced High Throughput Sequencing (HTS) datasets and would like to learn how to get the most out of it. This course will provide a general introduction to the bioinformatic and statistical aspects of HTS data analysis, alternating theory and practice. The objective is to present the general concepts such as file formats, mapping of sequences, as well as, the specific procedures for each data type: DNA-Seq, RNA-Seq, Chip-Seq. An additional session will be devoted to the pathway and functional analysis for gene set issued from differential or other kind of analysis.
Knowledge of the basic Linux and R commands is highly recommended. Otherwise it is advisable to follow the introductory tutorial available on the course web page.
Agenda
*Cambios sin previo aviso
Day 1 (Introduction day)
| Hora | Actividad |
| 8:00 - 8:30 | Welcome / registration |
| 8:30 a.m - 12:30 p.m | Presentation of the course [T] Introduction to HTS/NGS technologies (Illumina, PacBio, NanoPore, etc.) [T] Some basics stats/probability concepts; Particularities of NGS sampling; underlying distributions, etc. [T] |
| 2:00 p.m - 6:00 p.m | Quality analysis (error and bias of different technologies, quality metrics in sequencing, etc) [T/P] Introduction to file formats (FASTA, SAM, BED, BAM...) [T/P] |
Day 2 (First HTS day)
| Hora | Actividad |
| 8:30 a.m - 12:30 p.m | Alignment and mapping and algorithms [T] Mapping against a reference - Mapping for cleaning contaminants [P] |
| 2:00 p.m - 6:00 p.m | Assembly [T/P] |
Day 3 (Second HTS day)
| Hora | Actividad |
| 8:30 a.m - 12:30 p.m | Transcriptomics: Expression quantification, differential analysis, splice variant analysis [T/P] |
| 2:00 p.m - 6:00 p.m | Epigenomics [T/P]: Quality control of IP enrichmnet, peak calling and introduction to differential analysis [T/P] |
Day 4 (Annotation and visualization day)
| Hora | Actividad |
| 8:30 a.m - 12:30 p.m | Pathway analysis and functional analysis [T/P] |
| 2:00 p.m - 6:00 p.m | Introduction to analysis in Galaxy Visualization of datasets in the genome browser IGV |
Costo
$50.000
Descuentos:
Estudiantes* y egresados - 10%: $ 45.000
Afiliados a SisAndes - 20%: $ 40.000
Descuentos para grupos mayores a 10 personas - 30%: $ 35.000
*Los estudiantes deben presentar carnet vigente para ingresar al taller
Fechay hora
Fecha: 27 de Noviembre - 30 de Noviembre
Hora: 8:00 a.m. a 5:00 p.m.
Lugar
Waira 2
Universidad de los Andes - Edificio Mario Laserna
Cra. 1 Este No. 19 A - 40
Postulación
Please fill the following form (in English) in order to apply for participation in the "Hands-on course on High Throughput Sequencing data analysis: Genomics, Transcriptomics, Epigenomics".
The selection will be confirmed on Monday November 20. The deadline for registration and payment is November 24. Remember that the course will be partially taught in English.
Instructores

Alejandro Reyes Muñoz
Profesor Asociado de la Universidad de los Andes

Claudia Chica
Senior Bioinformatician - C3BI - Institut Pasteur, Paris, Francia.
As a computational biologist I have been involved in various projects seeking to answer different biological questions. Those projects have allowed me to define my main research interest, namely the evolutionary study of the emergence, storage and modulation of information in biological systems assisted by computational methods.
During my research career I have acquired extensive experience in the analysis of sequence data at the DNA and protein level. I’m trained both in NGS bioinformatic protocols (ChIP-seq, ATAC-seq, RNA-seq, genome assembly) and fine detail sequence analysis. Most importantly, I have gained proficiency in the use of the statistical models that are at the basis of the quantitative analysis of low and high throughput sequence data.
Additionally, my experience as a lecturer and instructor has taught me that training researchers about the formal basis of bioinformatic methodologies is the key for a successful collaboration between wet and dry lab. Likewise, I have gained valuable skills by working within two international consortia (TARA Oceans project and TRANSNET): the ability to collaborate with multidisciplinary groups and to coordinate younger researchers.

Rachel Legendre
Research Engineer - C3BI - Institut Pasteur, Paris, Francia
Rachel Legendre is a bioinformatics engineer. She completed her master degree in apprenticeship for two years at INRA in Jouy-en-Josas in the Genetic Animal department. She was involved in a project aiming at the detection and the expression analysis of micro-RNA involved in an equine disease.
In 2012, she joined the Genomic, Structure and Translation Team at Paris-Sud (Paris XI) university. She worked principally on Ribosome Profiling data analysis, a new technique that allows to identify the position of the ribosome on the mRNA at the nucleotide level.
Since november 2015, she joined the Bioinformatics and Biostatistics HUB at Pasteur Institute and she’s detached to the Biomics Pole in CITECH, where she is in charge of the bioinformatics analyses for Transcriptomic and Epigenetic Platform.




 FOROS ISIS
FOROS ISIS 
















